


近日,华南肿瘤学国家重点实验室贾卫华教授牵头,联合香港大学、南京医科大学、新疆医科大学、苏州大学及中山市人民医院、广西梧州红会医院等多个合作者团队,共同完成了一项大型多中心鼻咽癌遗传易感性研究,创建了可用于鼻咽癌发病风险预测的多基因风险评分新体系,并首次基于大规模前瞻性筛查人群队列,依据多基因风险评分高效识别高危人群,精准指导哪些个体可以从筛查中获益及个体化筛查建议起始年龄,将该发病风险评分与常规EB病毒抗体联用,可大幅改善鼻咽癌的筛查效能。该研究于4月12日在线发表于《自然通讯》(Nature communications),是中国学者针对我国华南地区特色高发疾病鼻咽癌防控领域的又一重要贡献。

高发区鼻咽癌疾病负担较重,防控手段有待改进
鼻咽癌,又称“广东癌”,在世界大部分地区均较为罕见,却唯独高发于我国华南及东南亚地区。据全球癌症统计数据显示,我国新发鼻咽癌病例约占全球的50%。近年来,随着诊疗技术的快速发展,对于早期鼻咽癌患者,其五年生存率高达90%以上。但由于鼻咽癌原发病灶较为隐匿、早期症状不明显等特点,约80%的患者发现时即为中晚期,病死率较高,对高发区居民造成沉重的疾病负担,且由于鼻咽癌高发于青壮年男性,对经济和社会均产生极大影响。
根据《癌症早诊早治项目技术方案》,建议对高发区30-59岁居民开展基于EB病毒抗体的筛查检测。目前我国有关鼻咽癌筛查的研究取得了阶段性进展,通过筛查极大提高了早诊率,降低了死亡率。但现有筛查方案有两个急待解决的问题:第一,现有EB病毒抗体检测方法虽具有较高的敏感性,但是特异性不足,阳性预测值<5%,即超过95%的阳性个体为假阳性,导致大量抗体阳性个人承受较大心理负担;第二,现行筛查方案无针对不同风险级别人群定制个体化的筛查策略。精准的癌症筛查策略应在综合考虑遗传、环境等多种危险因素的基础上,筛选出癌症的不同风险人群,然后针对不同风险人群制定切实有效的筛查和早诊、早治方案。因此,急需探究新方案来改善现有的筛查策略,提高筛查效率。
新一代鼻咽癌发病风险预测芯片可高效识别高危人群
研究团队针对该问题进行了长期的系统研究,组建了中国鼻咽癌遗传研究协作组。为了建立更为完整的遗传易感图谱,该协作组包括了来自鼻咽癌高、低发区的多个研究人群,利用多中心丰富的鼻咽癌样本资源、基因数据库及队列随访系统,开展了迄今为止全球最大的鼻咽癌全基因组关联研究,通过对约1万例个体全基因组的单核苷酸多态性位点(约500万个位点)进行系统分析,并经过两阶段独立人群验证,新发现了6个鼻咽癌遗传易感位点;在综合评估风险预测准确性和检测简便性的基础上,结合中肿鼻咽癌研究团队前期发现的遗传位点,构建了包含12个遗传位点的多基因风险评分(Polygenic risk score,PRS)模型,开发了新一代鼻咽癌发病风险预测芯片及软件【专利号:202011501223.8】。该风险预测体系可高效富集高危人群,且在低发区人群中也得到了很好验证,表现出较为一致且显著的剂量效应关系。
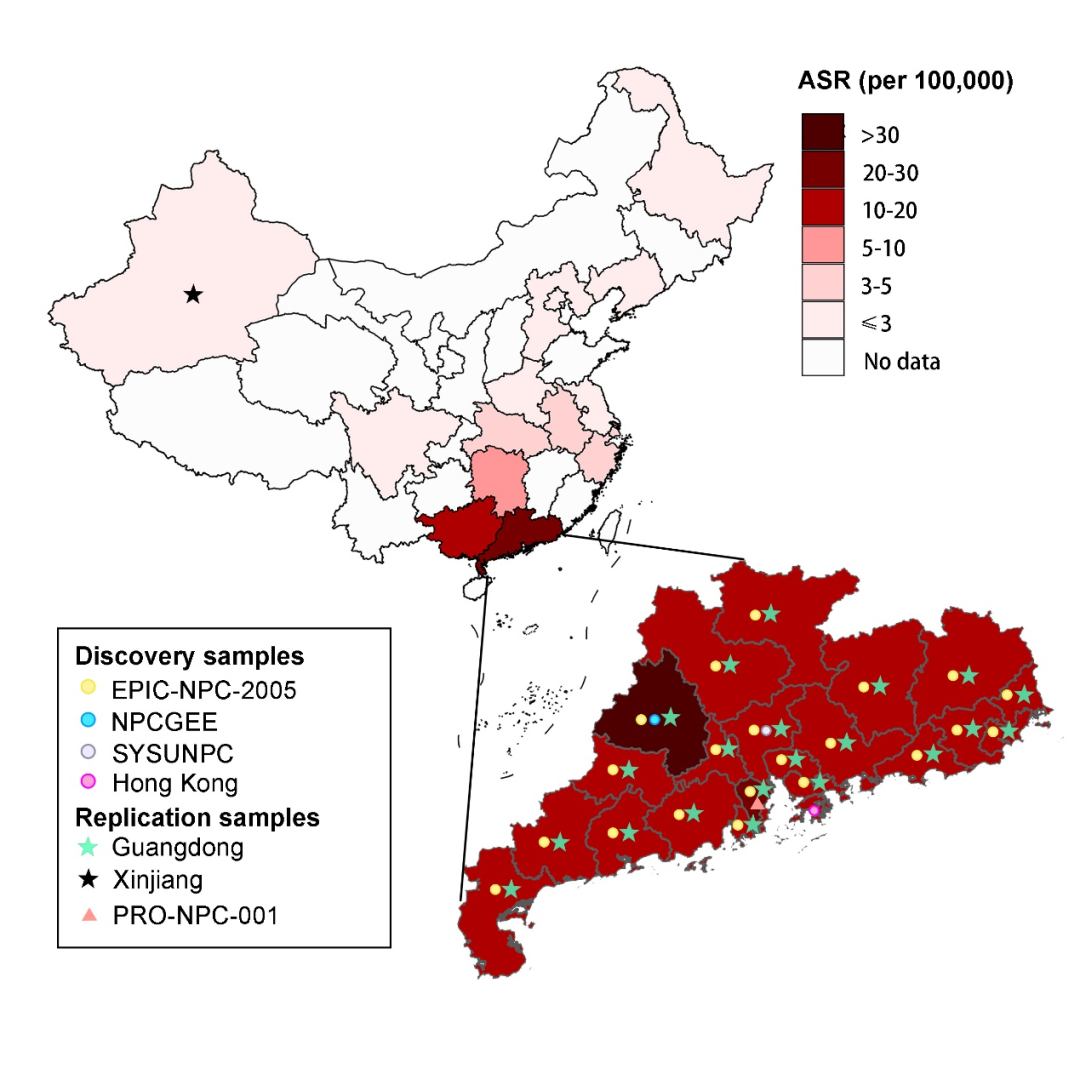
鼻咽癌综合风险评估新方案具有良好的应用潜力
前瞻性队列的研究设计是基于自然、真实的人群发病分布,可对发病风险进行客观、准确的评价,为研究提供高级别流行病学证据。团队首次基于包含约3万例人群近10年随访的中山市自然人群前瞻性筛查队列,对该PRS在鼻咽癌筛查中的效用进行系统评估:
(1)PRS可精准识别哪些个体可以从筛查中获益:联合现有的EB病毒抗体筛查,对于PRS较低的初筛抗体阳性者(PRS≤20%),平均筛查40个阳性者才发现1例鼻咽癌患者,而对于PRS较高者(PRS≥95%),平均筛查8个阳性者即可发现1例鼻咽癌患者。这表明,新建立的PRS既可以帮助识别低风险个体减少不必要的临床筛查,降低筛查假阳性率,减轻人群的心理负担;又能富集高风险个体,指导其积极开展临床筛查,提高筛查依从性,进而改善筛查效率。
(2)PRS有望对EB病毒抗体漏诊的个体进行补充:EB病毒抗体具有一定的动态波动性,部分初筛抗体阴性的个体后续也可能发展为鼻咽癌。团队发现,绝大部分漏诊者其PRS均处于较高的水平,如超过90%的漏诊者其PRS处于较高风险值(PRS≥50%),更有超过40%的漏诊者其PRS处于极高风险值水平(PRS≥90%)。PRS可以较好地对由于EB病毒抗体漏诊的个体进行风险预警,即对抗体检测的假阴性结果进行互补,对于这些漏诊者,如能及时给予PRS风险检测,将其判定为高风险者,将有望大大提高该部分漏诊者的早诊率。
(3)PRS为个体化精准筛查提供指导建议:根据推荐,建议高发区居民可从30岁开始进行鼻咽癌筛查。为提高筛查的成本效益,以男性为例,团队发现对于PRS≥90%的男性,由于其发病风险较高,其建议筛查起始年龄应提早到23岁;而对于PRS≤10%的男性,由于其发病风险较低,其筛查起始年龄可延迟到41岁,即对于携带不同PRS风险的个体,其建议筛查起始年龄存在个体化差异。
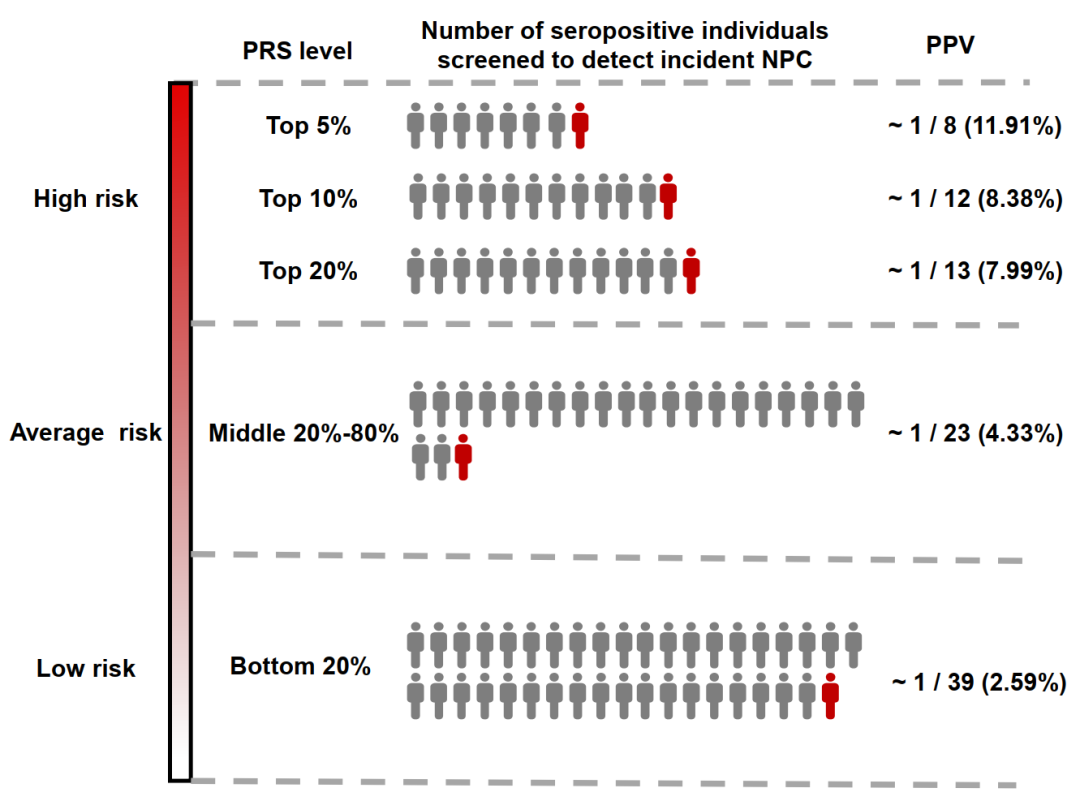
鼻咽癌筛查的阳性预测值(PPV)随多基因风险评分值(PRS)的增加而增加
PRS代表了一种个性化的遗传评估,它可以在任意年龄段检测,检测方法便捷,成本低廉。每个个体的PRS终生稳定,不易受时间、环境、生活行为习惯等干扰,只需一次检测,即可为指导个体是否需要筛查以及何时开始筛查等临床决策提供参考依据。该研究结果有望为我国高发区鼻咽癌高危人群的确定、发病风险预测、遗传咨询、筛查方案制定、甚至将来的预防型治疗提供科学依据。
值得提及的是,中大鼻咽癌团队前期就EB病毒亚型的研究提示在高发区存在特定的EB病毒高危亚型(Nature Genetics,2019;Virus Evolution,2021),该高危亚型在高发区健康人群中的感染率约为50%,但其在鼻咽癌患者中的感染率高达80%以上。同时,高危亚型在鼻咽癌低发区如中国北方人群感染率低于5%,在其他低发国家如欧洲和非洲等地则更为罕见,提示EB病毒高危亚型感染是鼻咽癌在华南地区高发的另一主要原因。
此项研究中,贾卫华教授、林大庆教授(香港大学)、叶为民教授(福建医科大学/瑞典卡罗琳斯卡医学院)、沈洪兵教授(南京医科大学)为共同通讯作者;何永巧博士、王曈旻博士、季明芳教授(中山市人民医院)、麦智明博士(香港大学)、汤敏中博士(广西梧州红会医院)、王若峥教授(新疆医科大学)、周翊峰教授(苏州大学)为共同第一作者。
本研究得到科技部国家重点研发计划、国家自然科学基金和广州市科技计划重点项目的支持,感谢Hans-Olov Adami(瑞典卡罗琳斯卡医学院/哈佛大学)、Jianjun Liu (新加坡基因研究所)、Maria Li Lung(香港大学)、Ellen T. Chang(美国斯坦福癌症研究所)、Zisis Kozlakidis(IARC国际癌症研究机构)对本研究提供的帮助,感谢本研究人群的所有参与者为科学研究做出的贡献。
研究者简介
最后通讯作者:贾卫华

中山大学肿瘤防治中心/华南肿瘤学国家重点实验室教授、博士生导师,华南肿瘤学国家重点实验室PI,肿瘤资源库负责人,致力于肿瘤个体化发病风险预测、早期诊断及筛查、肿瘤预后标志物的基础及转化应用研究。领导组建广东大型前瞻性自然人群队列、肿瘤临床生物样本库及家族性鼻咽癌研究队列。发展基于液体活检技术、基因组、转录组、微生态组等多组学的实验技术及高通量分析方法,用于肿瘤病因、早期干预及精准治疗的研究,积极推动成果转化。2013年获得国家自然科学基金杰出青年基金,2014年获得第十一届“中国青年女科学家”称号,2014年被聘为广东省“珠江学者”特聘教授,2016年入选国家级高层次人才。在国际著名杂志Nature Genetic,Nature communications,JNCI,Cancer Res等发表高水平论文百余篇。
现任中华预防医学会肿瘤预防与防控专业委员会副主任委员,中国抗癌协会肿瘤流行病学专业委员会副主任委员,中国医药生物技术协会组织生物样本库分会副主任委员,全国生物样本标准化技术委员会副主任委员。主持国家重点研发计划,国家自然科学基金重大国际合作项目,组织间国际合作项目等多项国家重大、重点项目资助。研究成果获国家自然科学奖二等奖、国家科技进步奖二等奖等多项奖励。
第一作者:何永巧

中山大学肿瘤防治中心/华南肿瘤学国家重点实验室特聘副研究员,致力于肿瘤遗传及基因组学,肿瘤分子流行病学等研究,作为主要负责人组建并承担广东大型前瞻性自然人群队列、中国南北方多中心研究队列及其生物样本资源库的建设。研究工作围绕肿瘤高发病因,从遗传易感、病毒感染、环境因素等多角度开展系统研究,为肿瘤病因、早期干预、精准防控及相关卫生决策的制定提供了重要的支持证据。作为课题负责人主持国家自然科学基金项目1项,作为任务负责人承担国家重点研发计划2项,近5年以第一/共同第一作者在Nature communications, Lancet Respir Med,Ebiomedicine,J Infect Dis,Cancer Biol Med等期刊发表多篇学术论文,申请及获批多项国家发明专利及软件著作权。
参考文献:1.Ji MF, Sheng W, Cheng WM, et al. Incidence and mortality of nasopharyngeal carcinoma: interim analysis of a cluster randomized controlled screening trial (PRO-NPC-001) in southern China. Ann Oncol 2019;30(10):1630-1637.2.Bei JX, Li Y, Jia WH, et al. A genome-wide association study of nasopharyngeal carcinoma identifies three new susceptibility loci. Nat Genet 2010;42(7):599-603.3.Xu M, Yao Y, Chen H, et al. Genome sequencing analysis identifies Epstein-Barr virus subtypes associated with high risk of nasopharyngeal carcinoma. Nat Genet 2019;51(7):1131-1136.4.Xue WQ, Wang TM, Huang JW, et al. A comprehensive analysis of genetic diversity of EBV reveals potential high-risk subtypes associated with nasopharyngeal carcinoma in China. Virus Evol 2021;7(1):veab010.